Mots-Clés
IS, annotation, machine learning
Description
Création d’une base de données d’éléments génétiques mobiles à partir de l’automatisation d’une annotation experte
L’automatisation et la systématisation des annotations expertes sont des enjeux cruciaux dans de nombreux domaines scientifiques. En bioinformatique, un problème classique concerne l’annotation des gènes, nécessaire aussi bien pour caractériser les organismes que pour en étudier les propriétés évolutives.
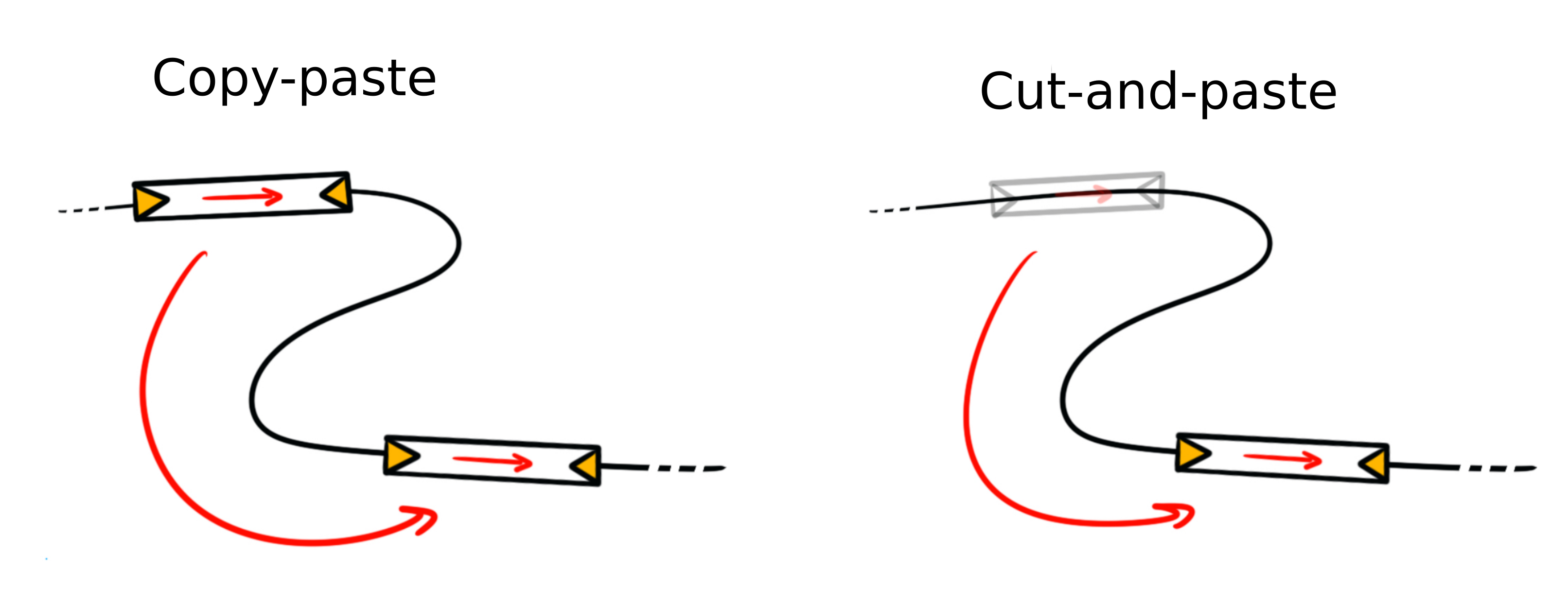
Dans ce stage, nous nous intéresserons à l’annotation des parties mobiles des génomes, c’est-à-dire des gènes dont la position et le nombre de copies évoluent très rapidement, posant des défis d’annotation particulièrement importants. Plus précisément, une partie des activités de recherche de notre équipe concerne la compréhension de la dynamique d’éléments génétiques nommés séquences d’insertion (IS) que l’on trouve chez les procaryotes (bactéries et archées). Les IS sont des séquences d’ADN capables de se déplacer et de se dupliquer de façon autonome d’une position à l’autre le long de la molécule d’ADN via des mécanismes de “copy-paste” ou “cut-and-paste”. Leur caractérisation est cruciale pour comprendre la dynamique évolutive des génomes.
Le stage consistera à développer un code python, sur la base d’arbres de décisions, afin d’automatiser une annotation experte fournie par des collègues du Centre de Biologie Intégrative (CBI) à Toulouse. Dans un premier temps, nous appliquerons ce code afin de raffiner le contenu d’une grande base de données d’IS disponible publiquement. Dans un deuxième temps, si le temps le permet, le travail consistera à mettre en place une approche d’apprentissage statistique (random forests) permettant de flexibiliser l’algorithme. Le code sera alors appliqué à la base de données publique afin de gagner en capacité d’annotation et d’améliorer la classification des IS.
Profil recherché : étudiante ou étudiant en maths / info avec un intérêt pour des problèmes appliqués à la biologie, une expérience de programmation en python et si possible d’analyse de données (par exemple, utilisation de pandas, matplotlib, scikit-learn).
Encadrement et environement scientifique : le stage aura lieu à Grenoble, au sein de l’équipe TrEE du laboratoire TIMC, au sein du groupe compbio@TrEE. Il sera encadré par Flora Gaudillière, Ivan Junier (expertise dynamique des génomes et des IS) et Nelle Varoquaux (expertise apprentissage statistique), dans le cadre d’une collaboration avec Patricia Siguier du CBI, Toulouse (experte en annotation manuelle des IS).
L’environnement grenoblois est riche en échanges scientifiques autour de la biologie computationnelle, avec entre autres l’initiative BiGre (Computational Biology in Grenoble). La communauté BiGre organise des séminaires scientifiques, des sessions de coworking et de formations, ainsi que des temps d’échange informels (avec pizza !).